Dalam beberapa dekade terakhir, ilmu genetika telah mengalami perkembangan pesat dengan munculnya berbagai teknologi yang memungkinkan para ilmuwan dan peneliti untuk mengurai kompleksitas genom manusia dan organisme lainnya. Salah satu teknologi yang menonjol dalam bidang ini adalah microarray, sebuah alat canggih yang digunakan untuk mengukur ekspresi gen, variasi genetik, serta pola metilasi DNA1.
Microarray adalah teknologi genomik memungkinkan kita untuk menganalisis ribuan hingga jutaan titik DNA dalam satu eksperimen. Teknologi ini memberikan gambaran menyeluruh tentang variasi genetika yang terdapat dalam sampel biologi, yang penting untuk berbagai aplikasi seperti deteksi penyakit, penemuan obat, dan penelitian genetika populasi2.

Ketika kita membandingkan dengan teknologi Next-Generation Sequencing (NGS), Teknologi NGS menawarkan kemampuan untuk membaca seluruh urutan genom atau segmen genom secara detail dan mendalam. NGS sangat berguna untuk menemukan mutasi langka atau baru yang belum diketahui sebelumnya. Namun, proses ini memerlukan kapasitas penyimpanan data yang besar dan biaya yang tinggi, terutama ketika menangani sejumlah besar sampel atau proyek berskala besar. Di sisi lain, teknologi microarray seperti Infinium dari Illumina lebih fokus pada variasi genetik yang sudah diketahui, seperti Single Nucleotide Polymorphisms (SNPs). Microarray memberikan keuntungan dalam hal efisiensi biaya dan waktu, terutama ketika tujuan penelitian adalah skrining genetik yang terarah pada varian yang umum dan telah dipahami dengan baik. Selain itu, microarray memungkinkan analisis genom secara luas dengan data yang lebih sedikit, membuatnya ideal untuk studi populasi besar atau penelitian epidemiologi yang membutuhkan penilaian genetik secara cepat dan tepat.
Bagaimana Teknologi Microarray Bekerja
Prinsip dasar dari teknologi microarray adalah kemampuannya untuk membaca variasi genetik spesifik yang sudah diketahui, seperti Single Nucleotide Polymorphisms (SNPs). Setiap titik pada microarray berfungsi seperti probe yang bisa mengenali urutan DNA tertentu dalam sampel yang dianalisis. Setelah sampel disiapkan dan ditempatkan pada chip microarray, berbagai proses biokimia dilakukan untuk memungkinkan probe mendeteksi dan berikatan dengan segmen DNA target.
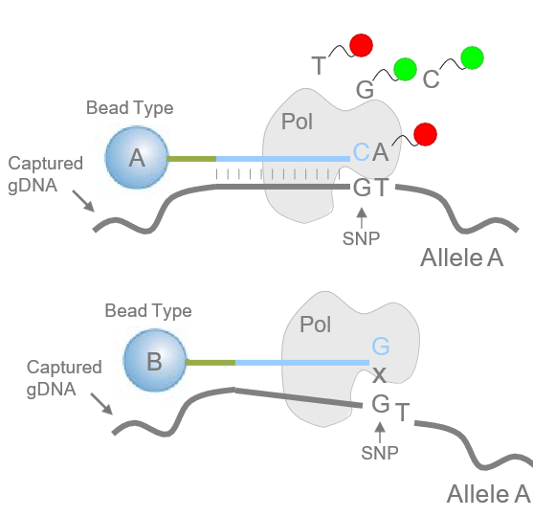
Sebagai hasilnya, microarray memberikan informasi tentang kehadiran atau ketidakhadiran variasi genetik pada ribuan lokasi di seluruh genom. Selain itu, microarray juga sering digunakan untuk studi asosiasi genom luas (Genome-Wide Association Studies atau GWAS), yang sangat penting dalam memahami bagaimana variasi genetik dapat memengaruhi risiko penyakit3.
Keunggulan dan Aplikasi Microarray dalam Studi Genom
Microarray memiliki sejumlah keunggulan yang membuatnya sangat berharga dalam penelitian genetika dan biologi molekuler. Di antara kelebihannya adalah biaya yang lebih rendah dan waktu yang lebih singkat dibandingkan dengan teknologi pengurutan generasi berikutnya (Next-Generation Sequencing, NGS), terutama ketika hanya variasi genetik spesifik yang ingin diteliti.
Aplikasi utama microarray meliputi:
- Genotyping: Menentukan profil genetika individu, termasuk untuk riset genetika populasi dan identifikasi variasi genetik terkait penyakit dan sifat lainnya4.
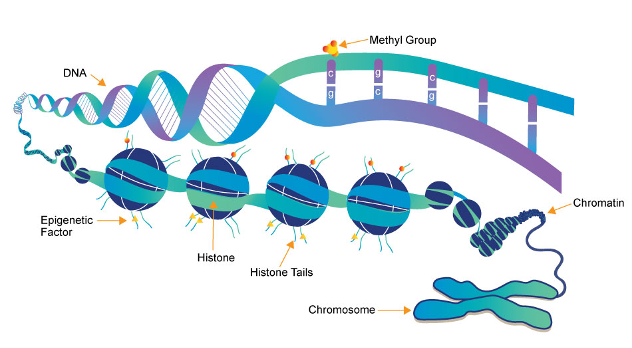
- Epigenetik: Studi metilasi DNA untuk memahami bagaimana faktor lingkungan dapat mengubah ekspresi gen tanpa memengaruhi urutan DNA5.
- Cytogenetik: Digunakan dalam penelitian kelainan kromosom seperti pada diagnosis kanker dan gangguan perkembangan6-7.
Teknologi Microarray Illumina: Infinium Assay dan iScan System
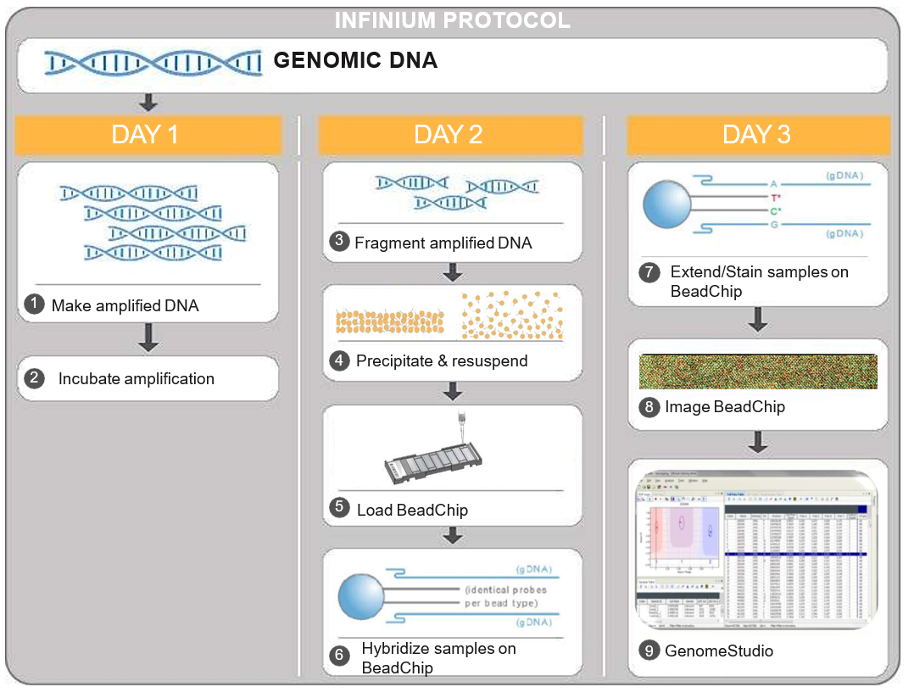
Pada titik ini, kami memperkenalkan teknologi microarray yang dikembangkan oleh Illumina, yang telah menjadi salah satu standar industri untuk penelitian genetika modern. Salah satu inovasi utama Illumina adalah Infinium Assay, yang menawarkan beberapa keunggulan utama, seperti presisi tinggi, reproduktibilitas yang sangat baik, dan kemampuan untuk mengolah berbagai tipe sampel biologis, termasuk darah, saliva, dan jaringan FFPE (formalin-fixed paraffin-embedded)8.

Teknologi Microarray Illumina menggunakan Infinium Assay dan iScan System, yang memungkinkan analisis genetik presisi tinggi dengan kompatibilitas untuk berbagai jenis sampel, termasuk darah, saliva, dan jaringan FFPE.
Dengan iScan System, Illumina menawarkan sistem pemindaian throughput tinggi yang dapat memproses ribuan sampel per minggu. Teknologi ini memungkinkan efisiensi yang lebih besar dalam analisis genetik, dengan kemampuan untuk mengotomatiskan banyak proses melalui AutoLoader 2.x, memberikan hasil yang lebih cepat dengan waktu pemrosesan minimal9.
Infinium vs Next-Generation Sequencing (NGS): Kapan Memilih Microarray?
Next-Generation Sequencing (NGS) dan Microarray adalah dua teknologi utama dalam analisis genom, masing-masing dengan keunggulan unik. NGS memberikan resolusi yang tinggi dengan membaca keseluruhan genom atau segmen besar dari DNA, yang membuatnya sangat bermanfaat untuk mendeteksi mutasi langka atau baru. Namun, teknologi ini membutuhkan lebih banyak data, kapasitas penyimpanan besar, dan biaya yang tinggi dalam pemrosesan dan analisisnya. Oleh karena itu, NGS biasanya lebih cocok untuk studi eksploratif yang memerlukan data komprehensif pada level genom penuh.
Sebaliknya, teknologi Microarray, seperti Infinium dari Illumina, dirancang untuk analisis varian genetik yang sudah diketahui, seperti Single Nucleotide Polymorphisms (SNPs). Dengan fokus pada variasi yang ditargetkan, Microarray lebih hemat biaya dan efisien dalam waktu. Hal ini menjadikannya ideal untuk aplikasi skala besar, seperti genotipe populasi dan studi epidemiologi, di mana hanya data pada titik-titik spesifik di seluruh genom yang diperlukan.
Teknologi Microarray juga memungkinkan kita untuk mengamati “gambaran keseluruhan” genom dengan data yang lebih ringkas dan lebih mudah diatur. Pada saat yang sama, Microarray dapat memuat hingga jutaan marker SNP, memberikan gambaran menyeluruh dari variasi genetik tanpa memerlukan data berlebihan seperti NGS10.
Aplikasi Khusus: Genotyping Populasi, Cytogenetik dan Epigenetik, serta Agrigenomik
Teknologi Infinium mendukung aplikasi genotyping dalam skala besar, yang sangat berguna untuk penelitian populasi genetik. Salah satu contohnya adalah studi Genome-Wide Association Studies (GWAS), yang memindai SNP di seluruh genom ribuan individu untuk mengidentifikasi variasi genetik terkait risiko penyakit kompleks, seperti asma, kanker, dan diabetes. Untuk aplikasi ini, Infinium Global Screening Array (GSA) v3 dan Infinium Global Diversity Array (GDA) menjadi pilihan utama, keduanya menawarkan cakupan varian yang luas dengan lebih dari 650.000 dan 1,9 juta marker, masing-masing. GDA bahkan dilengkapi dengan opsi Polygenic Risk Score (PRS) yang membantu dalam pengembangan skor risiko poligenik, yang sangat relevan untuk memprediksi kecenderungan genetik pada populasi besar11.
Infinium Assay juga memainkan peran penting dalam analisis metilasi DNA, yang merupakan perubahan epigenetik yang mengatur ekspresi gen tanpa memengaruhi urutan DNA. Salah satu pendekatan penelitian yang populer adalah Epigenome-Wide Association Studies (EWAS), yang digunakan untuk mengidentifikasi pola metilasi yang berbeda antara kelompok kasus dan kontrol. Studi ini telah digunakan untuk mengkaji dampak epigenetik terhadap risiko penyakit dan paparan lingkungan, seperti asma, efek merokok atau perubahan akibat penuaan12-13. Untuk studi epigenetik yang luas ini, Infinium MethylationEPIC v2.0 adalah produk andalan, menawarkan cakupan lebih dari 935.000 situs CpG, menjadikannya alat penting dalam penelitian epigenetik modern14.
iScan System dari Illumina, bersama Infinium Arrays, dapat mendeteksi variasi jumlah salinan DNA atau Copy Number Variants (CNV), yang penting dalam aplikasi cytogenetik dan diagnostik kanker. Aplikasi ini mencakup deteksi aneuploidi dan variasi kromosom pada kanker darah (leukemia dan limfoma) serta tumor padat. Infinium Global Screening Array (GSA) with Cytogenetics dan Global Diversity Array (GDA) with Cytogenetics mendukung aplikasi ini dengan tambahan hingga 160.000 marker yang relevan untuk penelitian klinis, memberikan cakupan yang lebih dalam pada lebih dari 4.800 gen yang terkait dengan penyakit. Teknologi ini memungkinkan laboratorium untuk memperoleh data akurat dalam analisis perubahan kromosom yang berkaitan dengan diagnosis kanker dan gangguan genetik lainnya.
Teknologi Infinium dari Illumina juga mendukung aplikasi agrigenomik dengan berbagai produk spesifik untuk hewan dan tanaman. Contoh produk ini termasuk BovineSNP50 v3 untuk sapi, OvineSNP50K v3 untuk domba, dan WheatBarley40k Array untuk tanaman gandum dan barley15. Selain itu, Illumina menyediakan opsi kustomisasi melalui Infinium HTS iSelect Custom Beadchips, yang memungkinkan peneliti untuk mendesain microarray sesuai kebutuhan spesifik untuk berbagai spesies. Produk ini sangat ideal untuk aplikasi seperti pemetaan sifat kompleks, verifikasi keturunan, dan seleksi genomik dalam program pemuliaan16. Dengan pilihan ini, teknologi Infinium mendukung peningkatan produktivitas dan keberagaman genetik dalam penelitian dan pengembangan agrikultur.
Teknologi Infinium tidak hanya mendukung aplikasi yang sudah dirancang khusus untuk studi genotyping, epigenetik, dan cytogenetik, tetapi juga memungkinkan penambahan konten custom. Pengguna dapat menambahkan konten kustom untuk memenuhi kebutuhan penelitian tertentu, seperti pada studi agrigenomik atau genomik non-manusia. Beberapa booster, seperti Neuro Booster untuk penyakit neurodegeneratif, Confluence Booster untuk kanker payudara, dan Multi-Disease Booster untuk berbagai penyakit kompleks, dirancang agar dapat ditambahkan ke Global Screening Array (GSA) atau Global Diversity Array (GDA) sesuai kebutuhan17. Dengan fleksibilitas ini, peneliti dapat mengadaptasi array untuk berbagai studi spesifik dan memanfaatkan potensi teknologi Infinium secara optimal.
Kesimpulan
Teknologi microarray, terutama yang dikembangkan oleh Illumina, menawarkan solusi yang kuat dan efisien untuk riset genetika, baik dalam skala kecil maupun besar. Dengan keunggulannya dalam hal presisi, kecepatan, dan skalabilitas, microarray tetap menjadi pilihan unggulan dalam berbagai aplikasi bioteknologi, dari penelitian dasar hingga pengembangan diagnostik dan terapi. Illumina dengan teknologi Infinium dan iScan System, memimpin dalam menyediakan platform yang dapat diandalkan dan efisien untuk kebutuhan genotiping, cytogenetik, dan epigenetik di era biologi molekuler modern.
Referensi
- Govindarajan R, Duraiyan J, Kaliyappan K, Palanisamy M. Microarray and its applications. J Pharm Bioallied Sci. 2012 Aug;4(Suppl 2): S310-2. doi:10.4103/0975-7406.100283. PMID: 23066278; PMCID: PMC3467903
- Gunderson KL, Steemers FJ, Lee G, Mendoza LG, Chee MS. A genome-wide scalable SNP genotyping assay using microarray technology. Nat Genet. 2005 May;37(5):549-54. doi:10.1038/ng1547. Epub 2005 Apr 17. PMID: 15838508
- Butler, H., Ragoussis, J. (2008). BeadArray-Based Genotyping. In: Starkey, M., Elaswarapu, R. (eds) Genomics Protocols. Methods in Molecular Biology™, vol 439. Humana Press. https://doi.org/10.1007/978-1-59745-188-8_4
- Steemers, F.J. and Gunderson, K.L. (2007), Whole genome genotyping technologies on the BeadArray™ platform. Biotechnology Journal, 2: 41-49. https://doi.org/10.1002/biot.200600213
- Bennett DA, Yu L, Yang J, Srivastava GP, Aubin C, De Jager PL. Epigenomics of Alzheimer’s disease. Transl Res. 2015 Jan;165(1):200-20. doi: 10.1016/j.trsl.2014.05.006. Epub 2014 May 16. PMID: 24905038; PMCID: PMC4233194.
- Esther H. Lips, Jan Willem F. Dierssen, Ronald van Eijk, Jan Oosting, Paul H.C. Eilers, Rob A.E.M. Tollenaar, Eelco J. de Graaf, Ruben van’t Slot, Cisca Wijmenga, Hans Morreau, Tom van Wezel; Reliable High-Throughput Genotyping and Loss-of-Heterozygosity Detection in Formalin-Fixed, Paraffin-Embedded Tumors Using Single Nucleotide Polymorphism Arrays. Cancer Res 15 November 2005; 65 (22): 10188–10191. https://doi.org/10.1158/0008-5472.CAN-05-2486
- Fung, Hon-Chung et al. (2006). Genome-wide genotyping in Parkinson’s disease and neurologically normal controls: first stage analysis and public release of data. The Lancet Neurology, Volume 5, Issue 11, 911 – 916
- Steemers, F. J., & Gunderson1,2, K. L. (2005). Illumina, Inc. Pharmacogenomics, 6(7), 777–782. doi:10.2217/14622416.6.7.777
- Adler, A. J., Wiley, G. B., Gaffney, P. M. Infinium Assay for Large-scale SNP Genotyping Applications. J. Vis. Exp. (81), e50683, doi:10.3791/50683 (2013).
- Seong Woon Roh, Guy C.J. Abell, Kyoung-Ho Kim, Young-Do Nam, Jin-Woo Bae. (2010). Comparing microarrays and next-generation sequencing technologies for microbial ecology research, Trends in Biotechnology, Volume 28, Issue 6, Pages 291-299. ISSN 0167-7799 https://doi.org/10.1016/j.tibtech.2010.03.001.
- Kaur, R.P., Shafi, G., Benipal, R.P.S. et al. Frequency of pathogenic germline mutations in cancer susceptibility genes in breast cancer patients. Med Oncol 35, 81 (2018). https://doi.org/10.1007/s12032-018-1143-2
- Cardenas, A., Sordillo, J.E., Rifas-Shiman, S.L. et al. The nasal methylome as a biomarker of asthma and airway inflammation in children. Nat Commun 10, 3095 (2019). https://doi.org/10.1038/s41467-019-11058-3
- Christiansen, C., Castillo-Fernandez, J.E., Domingo-Relloso, A. et al. Novel DNA methylation signatures of tobacco smoking with trans-ethnic effects. Clin Epigenet 13, 36 (2021). https://doi.org/10.1186/s13148-021-01018-4
- Pidsley, R., Zotenko, E., Peters, T.J. et al. Critical evaluation of the Illumina MethylationEPIC BeadChip microarray for whole-genome DNA methylation profiling. Genome Biol 17, 208 (2016). https://doi.org/10.1186/s13059-016-1066-1
- Tsai, HY., Janss, L.L., Andersen, J.R. et al. Genomic prediction and GWAS of yield, quality and disease-related traits in spring barley and winter wheat. Sci Rep 10, 3347 (2020). https://doi.org/10.1038/s41598-020-60203-2
- Haslin E, Pettigrew EJ, Hickson RE, Kenyon PR, Gedye KR, Lopez-Villalobos N, Jayawardana JMDR, Morris ST, Blair HT. Genome-Wide Association Studies of Live Weight at First Breeding at Eight Months of Age and Pregnancy Status of Ewe Lambs. Genes. 2023; 14(4):805. https://doi.org/10.3390/genes14040805
- Suratannon N, van Wijck RTA, Broer L, Xue L, van Meurs JBJ, Barendregt BH, van der Burg M, Dik WA, Chatchatee P, Langerak AW, Swagemakers SMA, Goos JAC, Mathijssen IMJ, Dalm VASH, Suphapeetiporn K, Heezen KC, Drabwell J, Uitterlinden AG, van der Spek PJ, van Hagen PM and the South East Asia Primary Immunodeficiencies (SEAPID) Consortium (2020) Rapid Low-Cost Microarray-Based Genotyping for Genetic Screening in Primary Immunodeficiency. Front. Immunol. 11:614. doi: 10.3389/fimmu.2020.00614
