Apa itu Next Generation Sequencing?
Next-Generation Sequencing (NGS) adalah teknologi modern dalam analisis genom yang mampu menghasilkan data urutan DNA dalam jumlah besar secara cepat dan efisien. Teknologi NGS memungkinkan analisis seluruh genom dengan biaya lebih rendah, menyediakan lebih dari satu miliar short reads dalam satu sesi instrumen1. Dengan kemampuan untuk menganalisis jutaan fragmen DNA secara paralel, NGS memungkinkan kita menjelajahi genom dengan kecepatan dan efisiensi yang tidak terbayangkan sebelumnya. Salah satu teknologi yang paling dominan dalam NGS adalah platform sequencing dengan pembacaan pendek (short-read), yang menawarkan keseimbangan sempurna antara akurasi tinggi dan throughput yang besar. Teknologi ini telah banyak digunakan dalam berbagai aplikasi penelitian, mulai dari sekuensing genom manusia hingga surveilans penyakit menular. Dalam artikel ini, kita akan mempelajari lebih jauh bagaimana NGS, terutama teknologi short-read, telah mengubah peta penelitian genetik di seluruh dunia, dan mengapa ia tetap menjadi pilihan utama bagi banyak ilmuwan dan klinisi.
Perbedaan Antara Long-read dan Short-read Sequencing
Next-generation sequencing (NGS) dapat dibedakan menjadi dua pendekatan utama berdasarkan panjang bacaannya: long-read sequencing dan short-read sequencing. Long-read sequencing memungkinkan pengurutan fragmen DNA yang panjangnya bisa mencapai lebih dari 10 kilobase (kb)2. Sebaliknya, short-read sequencing mengurutkan fragmen DNA yang lebih pendek, biasanya berkisar antara 150 hingga 300 pasangan basa (bp)3. Meski demikian, kedua teknologi ini menawarkan pendekatan berbeda dalam mengatasi tantangan genetika, tergantung pada tujuan spesifik penelitian. Beberapa brand yang menggunakan teknologi long-read sequencing adalah PacBio dan Oxford Nanopore, sementara Illumina adalah salah satu brand utama yang paling terkenal dalam menyediakan teknologi short-read sequencing.
Salah satu keuntungan utama dari long-read sequencing adalah kemampuannya untuk membaca fragmen DNA yang sangat panjang secara langsung, sehingga memudahkan dalam mendeteksi variasi structural yang kompleks dan region pengulangan (Genome-wide Repeat) dalam genom4. Teknologi ini juga sangat berguna untuk analisis haplotipe, di mana urutan alel dari satu orang dapat diidentifikasi secara akurat. Namun, long-read sequencing sering kali memiliki kelemahan dalam hal akurasi. Pada tahap awal pengembangannya, teknologi ini cenderung menghasilkan tingkat kesalahan yang lebih tinggi dibandingkan dengan short-read, meskipun kemajuan terbaru telah meningkatkan ketepatan datanya5.
Walaupun short-read sequencing memiliki keterbatasan dalam pengurutan fragmen panjang, seperti kesulitan dalam mengidentifikasi variasi struktural yang kompleks, teknologi ini tetap menjadi pilihan utama di banyak penelitian genetik karena beberapa kelebihan yang menonjol. Illumina, sebagai platform short-read paling populer, menawarkan akurasi yang sangat tinggi, bahkan mencapai kesalahan serendah 0,1% dalam pengurutan basa. Dengan inovasi terbaru seperti XLEAP SBS chemistry yang diterapkan pada instrumen baru seperti NovaSeq X, NextSeq 1000/2000, dan yang paling terbaru, MiSeq i100, Illumina berupaya untuk meningkatkan kecepatan dan efisiensi sequencing, mengatasi beberapa kelemahan yang ada pada teknologi sebelumnya6. Meskipun waktu yang dibutuhkan untuk sequencing bisa bervariasi, teknologi ini cenderung lebih hemat biaya, menjadikannya ideal untuk studi genomik berskala besar. Dengan kemampuan menghasilkan data dalam skala besar dan dukungan alat bioinformatika yang luas, short-read sequencing tetap mendominasi banyak aplikasi genomik di seluruh dunia.
Next-Generation Sequencing (NGS) telah merevolusi dunia genomik dengan memungkinkan analisis DNA secara paralel dan masif. Teknologi ini tidak hanya mempercepat proses sekuensing, tetapi juga meningkatkan akurasi data dengan metode yang lebih efisien dibandingkan teknik sekuensing generasi sebelumnya. Salah satu teknologi utama dalam NGS adalah metode Sequencing by Synthesis (SBS) dari Illumina, yang kini digunakan untuk menghasilkan lebih dari 90% data sekuensing di dunia.
Bagaimana Workflow dari NGS dengan Sistem Sequencing by Synthesis (SBS)?
Proses NGS dengan sistem SBS dibagi menjadi empat tahap utama: persiapan perpustakaan DNA (library preparation), klusterisasi (cluster generation), sekuensing, dan analisis data.
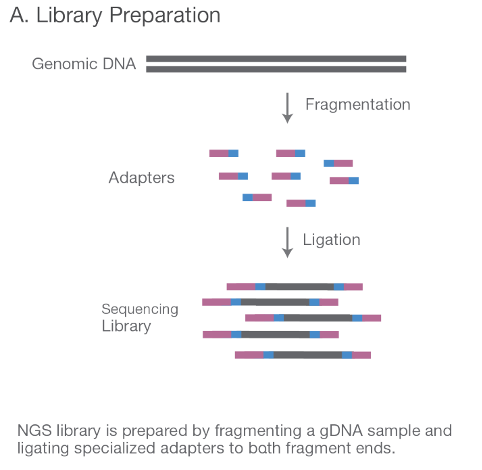
1. Library Preparation
Pertama, sampel DNA atau cDNA difragmentasi secara acak dan diberi adaptor pada kedua ujungnya. Fragmen ini kemudian di-amplifikasi dengan PCR dan dimurnikan untuk membentuk perpustakaan DNA yang siap digunakan7.
2. Klusterisasi
Dalam teknologi Next-Generation Sequencing (NGS), salah satu elemen kunci adalah flowcell. Flowcell adalah perangkat tempat reaksi sekuensing berlangsung di mesin sekuensing Illumina. Flowcell ini memiliki permukaan yang dilapisi dengan oligonukleotida yang berfungsi untuk mengikat fragmen DNA yang sudah dipersiapkan sebelumnya (tahap library preparation). Proses ini sangat penting karena memungkinkan ratusan juta fragmen DNA diurutkan secara paralel, menghasilkan data sekuens yang luar biasa besar dalam satu putaran sekuensing.
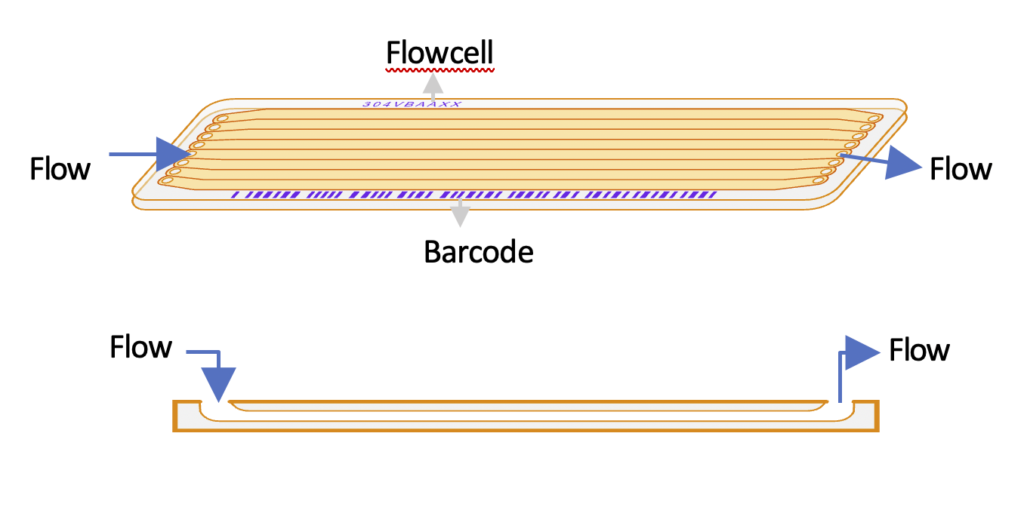
Klusterisasi (cluster generation) adalah langkah penting yang terjadi di dalam flowcell. Setelah fragmen DNA diikatkan ke permukaan flowcell, proses klusterisasi dimulai. Dalam proses ini, setiap fragmen DNA diperbanyak melalui reaksi amplifikasi secara isothermal bridge amplification. Fragmen DNA yang terikat pada oligonukleotida akan dibiakkan hingga terbentuk ribuan salinan yang identik di tempat yang sama, membentuk kluster. Setiap kluster mewakili satu molekul DNA yang diperbanyak, dan inilah yang akan diurutkan selama proses sekuensing. Klusterisasi terjadi langsung di dalam flowcell sebelum proses sekuensing dimulai.

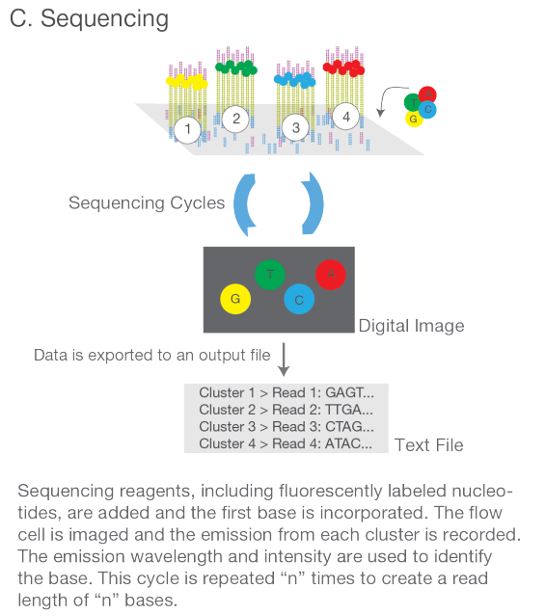
3. Proses Sekuensing
Illumina menggunakan metode yang disebut Sequencing by Synthesis (SBS), di mana nukleotida yang ditandai dengan pewarna fluoresen ditambahkan ke rantai DNA yang sedang tumbuh. Setiap nukleotida memiliki warna tertentu yang akan dideteksi oleh kamera saat ditambahkan. Proses ini dilakukan secara bersamaan pada jutaan kluster di flowcell, sehingga ribuan hingga miliaran basa DNA dapat dibaca dalam satu putaran sekuensing. Dalam metode ini, penting untuk memahami dua parameter utama: panjang baca (read length), yang menunjukkan jumlah basa yang dibaca dalam setiap siklus, dan kedalaman baca (read depth), yang mengacu pada jumlah pembacaan yang diperoleh dari satu sampel. Kedua parameter ini harus disesuaikan dengan tujuan eksperimen yang spesifik.
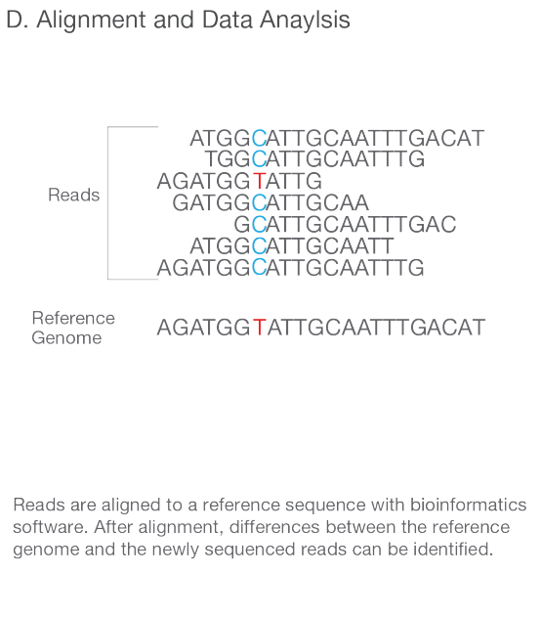
4. Analisis Data Sekuensing
Setelah proses sekuensing selesai, tahap berikutnya adalah analisis data. Setiap nukleotida yang dibaca oleh mesin Illumina dikonversi menjadi data mentah berupa urutan A, T, G, dan C, yang disimpan dalam format FASTQ. Data ini kemudian diproses lebih lanjut melalui berbagai alat bioinformatika untuk mengidentifikasi varian genetik atau untuk menginterpretasi hasil penelitian lainnya. Salah satu keuntungan utama dari NGS Illumina adalah kemampuannya untuk menangani analisis yang sangat kompleks menggunakan platform bioinformatika yang canggih seperti BaseSpace® Sequence Hub. Analisis ini memungkinkan ilmuwan untuk memahami pola ekspresi gen, mutasi, atau variasi struktural dalam sampel dengan tingkat kedalaman dan detail yang belum pernah ada sebelumnya.
Keunggulan Teknologi SBS: Akurasi Tinggi dan Keserbagunaan
SBS Illumina menggunakan terminator reversibel berbasis fluoresens, di mana setiap nukleotida yang terintegrasi akan menghasilkan sinyal fluoresen yang dapat dideteksi. Proses ini berlangsung dalam siklus yang terus menerus berlangsung, dengan semua jenis nukleotida terus dialirkan dalam setiap siklus, memastikan bahwa tidak ada bias dalam penggabungan basa. Hasilnya adalah data sekuensing yang sangat akurat dengan tingkat kesalahan yang sangat rendah, bahkan dalam pengulangan sekuens (repeated sequence) atau homopolimer yang sulit8,9.
Teknologi NGS dengan SBS dari Illumina menawarkan berbagai keunggulan yang tidak hanya mencakup akurasi dan skalabilitas, tetapi juga efisiensi waktu dan biaya. Dengan kemampuan untuk menyesuaikan jumlah pembacaan sekuens yang dihasilkan, para peneliti dapat mengoptimalkan eksperimen mereka untuk berbagai tujuan, dari penelitian genetik dasar hingga aplikasi klinis yang lebih kompleks. Sistem NGS Illumina juga didukung oleh alat bioinformatika terintegrasi seperti BaseSpace® Sequence Hub, yang memungkinkan analisis data dilakukan secara otomatis dan efisien.
Aplikasi Aplikasi di High-Throughput Sequencer


Teknologi High-Throughput sequencer seperti NovaSeq X dari Illumina memungkinkan sekuensing genom skala besar yang revolusioner dengan memanfaatkan kemajuan kimia dan analisis data. High-Throughput sequencer mampu menghasilkan throughput yang tinggi dengan resolusi yang sangat mendalam, cocok untuk studi genomik yang memerlukan analisis variabilitas genetik pada skala populasi besar, seperti Genome-wide Association Studies (GWAS). Aplikasi utamanya meliputi Whole Genome Sequencing (WGS) dan eksom untuk mendeteksi varian yang berhubungan dengan penyakit kompleks, termasuk kanker, penyakit genetik, dan penyakit langka dan Whole Exome Sequencing (WES) yang memungkinkan identifikasi mutasi yang lebih spesifik dengan cakupan wilayah eksom yang lebih dalam 10, 11.
Studi kanker sering menggunakan sekuensing DNA tumor vs DNA normal untuk membandingkan mutasi somatik dalam tumor dengan jaringan normal, yang membantu dalam identifikasi mutasi yang berperan sebagai driver mutations dalam perkembangan kanker12,13. Dalam studi penyakit genetik, NovaSeq X juga memungkinkan penggunaan metode multiomik, termasuk sekuensing RNA dan DNA, yang sangat efektif dalam mengungkap bagaimana mutasi genetik mempengaruhi ekspresi gen dan protein yang relevan dengan patologi penyakit14, 15.
Dalam studi epigenetik, teknik seperti ATAC-Seq dan Methylation Sequencing sering digunakan. ATAC-Seq (Assay for Transposase-Accessible Chromatin with high-throughput sequencing) digunakan untuk mengkaji aksesibilitas kromatin, yang memberikan informasi tentang regulasi genetik berdasarkan keterbukaan daerah DNA. Methylation sequencing digunakan untuk menganalisis pola metilasi DNA yang berperan penting dalam regulasi gen dan dapat membantu memahami mekanisme penyakit yang dipengaruhi oleh faktor epigenetik16-20.
Untuk analisis transkriptom, RNA-Seq (RNA sequencing) merupakan teknik utama yang digunakan untuk memetakan ekspresi gen pada tingkat RNA. RNA-Seq memungkinkan peneliti untuk melihat variasi dalam ekspresi gen, termasuk identifikasi isoform transkrip alternatif, serta mendeteksi fusi gen yang tidak terungkap dalam analisis DNA21. Single-cell RNA sequencing (scRNA-Seq) juga digunakan untuk memahami heterogenitas seluler, terutama dalam konteks studi imunologi dan kanker, di mana peneliti dapat mengidentifikasi perbedaan ekspresi gen antar sel secara individual22. 23.
Dalam profiling multiomik, NovaSeq X memungkinkan penggabungan berbagai jenis data molekuler, termasuk genomik, transkriptomik, dan proteomik. Integrasi berbagai omes ini memberikan gambaran yang lebih menyeluruh tentang mekanisme biologis yang kompleks24- d 26.
Aplikasi Benchtop Sequencer dalam Penelitian Skala Lebih Kecil


Untuk laboratorium dengan kebutuhan throughput lebih rendah, Illumina menawarkan benchtop sequencer seperti MiSeq i100 dan NextSeq, yang tetap menawarkan fleksibilitas dan presisi yang tinggi. Alat ini cocok untuk aplikasi seperti Small Whole-Genome Sequencing27 (genome virus atau bakteri), metagenomik mikroba, profiling sel Tunggal (Sinlge-Cell sequencing), dan epigenetik. Misalnya, sekuensing 16S rRNA adalah salah satu metode yang sering digunakan untuk mengkaji komunitas mikroba, memungkinkan peneliti untuk mengidentifikasi mikroorganisme yang sulit dikultur dan memahami keanekaragaman mikrobiota dalam berbagai lingkungan28,29.
Metagenomik shotgun juga sering digunakan dalam studi penyakit menular, di mana sekuensing mikroba dari sampel kompleks dapat membantu dalam mengidentifikasi patogen atau melacak penyebaran penyakit seperti wabah infeksi virus30. Dalam studi terkait keamanan pangan, sekuensing genom mikroba membantu dalam memetakan hubungan epidemiologis antara sumber makanan yang terkontaminasi dan wabah penyakit.
Sistem benchtop sequencer Illumina juga mendukung penelitian epigenetik, yang memungkinkan peneliti untuk mengungkap mekanisme regulasi genetik yang berperan dalam perkembangan penyakit31. Teknik seperti ATAC-Seq untuk studi aksesibilitas kromatin dan methylation sequencing untuk analisis metilasi DNA telah memanfaatkan sistem ini untuk memberikan wawasan mendalam tentang interaksi gen dan faktor lingkungan. Selain itu, kemampuan untuk mengintegrasikan multiomik dan profiling sel tunggal dengan alat seperti NextSeq 2000 memungkinkan peneliti untuk menganalisis ekspresi gen pada resolusi sel Tunggal (single-cell), yang sangat penting dalam penelitian imunologi, kanker, dan bidang lainnya.
Selain itu, targeted sequencing juga sering diterapkan, di mana panel gen atau wilayah genom tertentu dianalisis secara mendalam. Teknik ini ideal untuk studi klinis atau diagnostik genetik, terutama untuk deteksi mutasi yang terkait dengan penyakit, seperti panel gen kanker32. Targeted resequencing memungkinkan peneliti untuk fokus pada gen-gen spesifik yang relevan dengan kondisi medis tertentu, memberikan data yang sangat akurat tanpa perlu mengurutkan seluruh genom.
Untuk studi transkriptomik, benchtop sequencer dapat menggunakan RNA-Seq skala kecil, yang memberikan gambaran ekspresi gen dalam sampel tertentu. Ini sangat berguna dalam penelitian sel tunggal atau analisis ekspresi gen spesifik pada berbagai jenis jaringan.
Kesimpulan
Teknologi Next-Generation Sequencing (NGS) telah membawa perubahan revolusioner dalam dunia genomik, memungkinkan para peneliti untuk menganalisis DNA secara cepat dan akurat. Dengan platform seperti Illumina yang mendominasi penggunaan di berbagai aplikasi ilmiah, NGS memfasilitasi penelitian dalam skala besar dan memperluas pemahaman kita tentang genetika, penyakit kompleks, serta variasi genomik. Selain menawarkan akurasi tinggi dan throughput yang luar biasa, NGS juga menyediakan fleksibilitas bagi laboratorium dengan skala penelitian yang lebih kecil. Dengan demikian, NGS terus menjadi teknologi pilihan utama dalam bidang genetika dan biologi molekuler, berperan penting dalam penelitian klinis, penyakit genetik, dan berbagai studi multiomik lainnya
Referensi
- Shendure, J., Ji, H. Next-generation DNA sequencing. Nat Biotechnol 26, 1135–1145 (2008). https://doi.org/10.1038/nbt1486
- Taishan Hu, Nilesh Chitnis, Dimitri Monos, Anh Dinh. Next-generation sequencing technologies: An overview, Human Immunology. Volume 82, Issue 11, Pages 801-811 (2021). ISSN 0198-8859, https://doi.org/10.1016/j.humimm.2021.02.012
- T. Tucker, M. Marra, J.M. Friedman. massively parallel sequencing: the next big thing in genetic medicine. Am. J. Hum. Genet. 85, 142 (2009). 10.1016/j.ajhg.2009.06.022
- E.L. van Dijk, Y. Jaszczyszyn, D. Naquin, C. Thermes. The Third Revolution in Sequencing Technology. Trends Genet 34, 666 (2018). https://doi.org/10.1016/j.tig.2018.05.008
- Logsdon, G.A., Vollger, M.R. & Eichler, E.E. Long-read human genome sequencing and its applications. Nat Rev Genet 21, 597–614 (2020). https://doi.org/10.1038/s41576-020-0236-x
- Illumina. (n.d.). Understanding data quality and Q scores. Retrieved October 15, 2024, from https://sapac.illumina.com/science/genomics-research/articles/data-quality-q-scores.html
- llumina. Nextera DNA Library Preparation Kits data sheet. 2014. (www.illumina.com/documents/products/datasheets/datasheet_nextera_dna_sample_prep.pdf)
- Ross MG, Russ C, Costello M, et al. Characterizing and measuring bias in sequence data. Genome Biol. 2013;14(5):R51.
- Bentley DR, Balasubramanian S, Swerdlow HP, et al. Accurate whole human genome sequencing using reversible terminator chemistry. Nat. 2008;456(7218):53– 59
- Turro E, Astle WJ, Megy K, et al. Whole-genome sequencing of patients with rare diseases in a national health system. Nature. 2020;583(7814):96-102. doi:10.1038/s41586-020-2434-2
- Meienberg J, Bruggmann R, Oexle K, Matyas G. Clinical sequencing: is WGS the better WES? Hum Genet. 2016;135(3):359-362. doi:10.1007/s00439-015-1631-9
- Duncavage EJ, Bagg A, Hasserjian RP, et al. Genomic profiling for clinical decision making in myeloid neoplasms and acute leukemia. Blood. 2022;140(21):2228-2247. doi:10.1182/blood.2022015853
- Duncavage EJ, Schroeder MC, O’Laughlin M, et al. Genome Sequencing as an Alternative to Cytogenetic Analysis in Myeloid Cancers. N Engl J Med. 2021;384(10):924-935. doi:10.1056/NEJMoa2024534
- Colin E, Duffourd Y, Tisserant E, et al. OMIXCARE: OMICS technologies solved about 33% of the patients with heterogeneous rare neuro-developmental disorders and negative exome sequencing results and identified 13% additional candidate variants. Front Cell Dev Biol. 2022;10:1021785. doi:10.3389/fcell.2022.1021785
- Kamali Z, Keaton JM, Haghjooy Javanmard S, et al. Large-Scale Multi-Omics Studies Provide New Insights into Blood Pressure Regulation. Int J Mol Sci. 2022;23(14):7557. doi:10.3390/ijms23147557
- Lissa D, Robles AI. Methylation analyses in liquid biopsy. Transl Lung Cancer Res. 2016;5(5):492-504. doi:10.21037/tlcr.2016.10.03
- Laird PW. The power and the promise of DNA methylation markers. Nat Rev Cancer. 2003;3(4):253-266. doi:10.1038/nrc1045
- Huang J, Wang L. Cell-Free DNA Methylation Profiling Analysis-Technologies and Bioinformatics. Cancers. 2019;11(11):1741. doi:10.3390/cancers11111741
- Luo H, Wei W, Ye Z, Zheng J, Xu RH. Liquid Biopsy of Methylation Biomarkers in Cell-Free DNA. Trends Mol Med. 2021;27(5):482-500. doi:10.1016/j. molmed.2020.12.011
- Warton K, Lin V, Navin T, et al. Methylation-capture and Next-Generation Sequencing of free circulating DNA from human plasma. BMC Genomics. 2014;15(1):476. doi:10.1186/1471-2164-15-476
- Splinter K, Adams DR, Bacino CA, et al. Effect of Genetic Diagnosis on Patients with Previously Undiagnosed Disease. N Engl J Med. 2018;379(22):2131-2139. doi:10.1056/NEJMoa1714458
- Cao J, Spielmann M, Qiu X, et al. The single-cell transcriptional landscape of mammalian organogenesis. Nature. 2019;566(7745):496-502. doi:10.1038/s41586-019-0969-x
- Plasschaert LW, Žilionis R, Choo-Wing R, et al. A single-cell atlas of the airway epithelium reveals the CFTR-rich pulmonary ionocyte. Nature. 2018;560(7718):377-381. doi:10.1038/s41586-018-0394-6
- Colin E, Duffourd Y, Tisserant E, et al. OMIXCARE: OMICS technologies solved about 33% of the patients with heterogeneous rare neuro-developmental disorders and negative exome sequencing results and identified 13% additional candidate variants. Front Cell Dev Biol. 2022;10:1021785. doi:10.3389/fcell.2022.1021785
- Kamali Z, Keaton JM, Haghjooy Javanmard S, et al. Large-Scale Multi-Omics Studies Provide New Insights into Blood Pressure Regulation. Int J Mol Sci. 2022;23(14):7557. doi:10.3390/ijms23147557
- Lunke S, Bouffler SE, Patel CV, et al. Integrated multi-omics for rapid rare disease diagnosis on a national scale. Nat Med. 2023:1-11. doi:10.1038/s41591-023-02401-9
- Wensel CR, Pluznick JL, Salzberg SL, Sears CL. Next-generation sequencing: insights to advance clinical investigations of the microbiome. J Clin Invest. 2022;132(7):e154944. doi:10.1172/JCI154944
- Clarridge JE 3rd. Impact of 16S rRNA gene sequence analysis for identification of bacteria on clinical microbiology and infectious diseases. Clin Microbiol Rev. 2004;17(4):840-862. doi:10.1128/CMR.17.4.840-862.2004
- McDonald D, Hyde E, Debelius JW, et al. American Gut: an Open Platform for Citizen Science Microbiome Research. mSystems. 2018;3(3):e00031-18. doi:10.1128/mSystems.00031-18
- Isidro J, Borges V, Pinto M, et al. Phylogenomic characterization and signs of microevolution in the 2022 multi-country outbreak of monkeypox virus. Nat Med. 2022;28(8):1569-1572. doi:10.1038/s41591-022-01907-y
- Tiffon C. The Impact of Nutrition and Environmental Epigenetics on Human Health and Disease. Int J Mol Sci. 2018;19(11):3425. doi:10.3390/ijms19113425
- Kina BG, Topbas Selcuki NF, Bahat PY, et al. Whole exome sequencing reveals novel candidate variants for endometriosis utilizing multiple affected members in a single family. Mol Genet Genomic Med. 2024;12(1):e2312. doi:10.1002/mgg3.2312
- Ke M, Elshenawy B, Sheldon H, Arora A, Buffa FM. Single cell RNA-sequencing: A powerful yet still challenging technology to study cellular heterogeneity. Bioessays. 2022;44(11):e2200084. doi:10.1002/ bies.202200084
